据英国《自然》杂志16日发表的一项结构生物学最新研究,世界著名人工智能团队深度思维(DeepMind)描述了神经网络“阿尔法折叠2”能以就计算机方法而言前所未有的准确度,根据蛋白质的氨基酸序列预测其三维结构。
蛋白质折叠问题被认为是人类在21世纪需要解决的重要科学前沿问题之一。理解蛋白质的结构有助于确定蛋白质的功能,了解各种突变的作用。截至目前,约有10万个蛋白质的结构已经用实验方法得到了解析,但这在已经测序的数10亿计的蛋白质中只占了很小一部分。在50多年的时间里,研究人员一直尝试根据蛋白质的氨基酸序列预测其折叠而成的三维结构。然而,当前使用的计算方法准确度有限,实验方法对人力和时间的要求也非常高。
此次,深度思维首席科学家约翰·詹普尔、创始人兼首席执行官戴米斯·哈萨比斯及其团队描述了“阿尔法折叠2”——一个基于神经网络的新模型,其预测的蛋白质结构能达到原子水平的准确度。研究团队在2020年5月至7月举办的第14届“蛋白质结构预测关键评估”(CASP14)大赛中验证了这种方法。
CASP14比赛要求参赛团队根据蛋白质的氨基酸序列解析它们的结构。比赛用的蛋白质会先用实验方法解析出来,但具体结果不会公开。比赛中,“阿尔法折叠2”预测的大部分结构达到了空前的准确度,不仅与实验方法不相上下,还远超解析新蛋白质结构的其他方法。将实验方法得到的蛋白质结构叠加在“阿尔法折叠2”的结构上,组成蛋白质主链骨架的叠加原子之间的距离中位数(95%的覆盖率)为0.96埃(0.096纳米)。成绩排第二的方法只能达到2.8埃的准确度。
“阿尔法折叠2”的神经网络能在几分钟内预测出一个典型蛋白质的结构,还能预测较大蛋白质(比如一个含有2180个氨基酸、无同源结构的蛋白质)的结构。该模型能根据每个氨基酸对其预测可靠性进行精确预估,方便研究人员使用其预测结果。
研究团队认为,这一精准的预测算法可以让蛋白质结构解析技术跟上基因组革命的发展步伐。
戴米斯·哈萨比斯在一份声明中表示,他们将为科学共同体提供广泛、免费的获取途径且已迈出承诺的第一步——在《自然》期刊上分享“阿尔法折叠”的开源代码,并发表了系统的完整方法论,以期待看到该方法为科学界启发出其他新的研究方法。
总编辑圈点
我们之所以能说话、会思考、善学习、有情感,与人脑中860亿个神经元、几千亿个神经胶质细胞、100万亿个神经突触密切相关。但人脑的具体工作机制到底是怎样的?面纱仍在一步步揭开。这背后,离不开科研人员在脑科学领域点点滴滴的进步与突破。(记者张梦然)
 全球观察:端午节期间日均132.1万人次出入境 较去年增长约2.3倍 据国家移民管理局25日消息,2023年端午节期间全国边检机关共查验出入境
全球观察:端午节期间日均132.1万人次出入境 较去年增长约2.3倍 据国家移民管理局25日消息,2023年端午节期间全国边检机关共查验出入境
 汽车博主发表贬损极狐汽车言论 极狐要求永久删除 近日,北汽极狐公司针对知名汽车博主袁启聪在抖音和微博发布的涉及极狐
汽车博主发表贬损极狐汽车言论 极狐要求永久删除 近日,北汽极狐公司针对知名汽车博主袁启聪在抖音和微博发布的涉及极狐  天天视点!极限 极限有高有低……心理极限极高的人可能身体会先到极限而出问题。身体极
天天视点!极限 极限有高有低……心理极限极高的人可能身体会先到极限而出问题。身体极  世界热推荐:观众齐喊“开空调”!梁静茹沈阳演唱会遭遇尴尬一幕 近期高温预警,再加上演出现场人员爆满,昨天是满场,估计昨天观看演唱
世界热推荐:观众齐喊“开空调”!梁静茹沈阳演唱会遭遇尴尬一幕 近期高温预警,再加上演出现场人员爆满,昨天是满场,估计昨天观看演唱 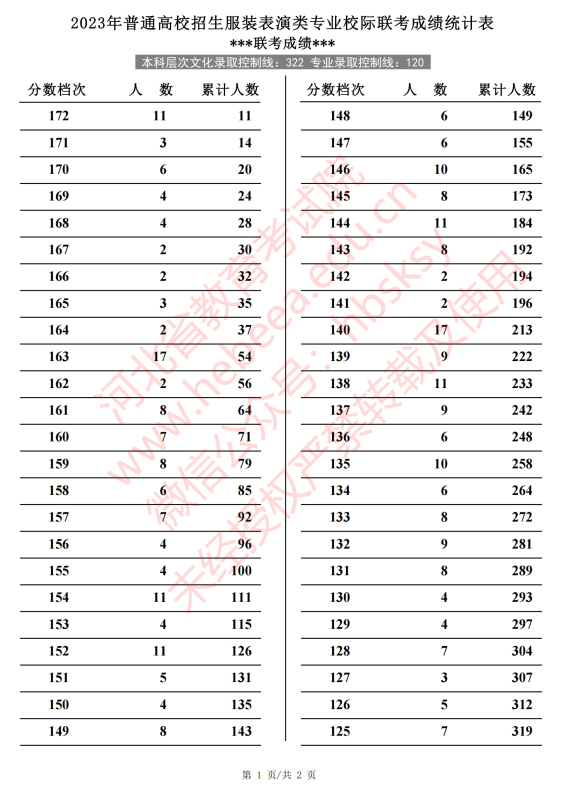 每日快看:2023年河北省普通高校招生服装表演类专业校际联考成绩统计表(专业成绩·综合成绩) 最新高考资讯、高考政策、考前准备、志愿填报、录取分数线等高考时间线
每日快看:2023年河北省普通高校招生服装表演类专业校际联考成绩统计表(专业成绩·综合成绩) 最新高考资讯、高考政策、考前准备、志愿填报、录取分数线等高考时间线  海口江东新区将建香港玉玲珑珠宝产业园 近日,记者从海口江东新区管理局获悉,位于海口江东新区离岸创新创业组
海口江东新区将建香港玉玲珑珠宝产业园 近日,记者从海口江东新区管理局获悉,位于海口江东新区离岸创新创业组